Кажется, нашел немного более простую функцию пригодности для использования с ГА. В первом своем сообщении я уже сделал небольшое упрощение, отказавшись от МНК, а на этот раз я предлагаю одну из сигм заменить на минимум:
![$$\sum_{i=1}^n\min_{j\in[1;\ k]}\|x_j-y_i\|\to\min,$$ $$\sum_{i=1}^n\min_{j\in[1;\ k]}\|x_j-y_i\|\to\min,$$](https://dxdy-03.korotkov.co.uk/f/a/1/e/a1e469d09e5a9496923fa25ea055de1282.png)
где

-- затравки кластеров,
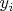
-- данные. Интуитивно чувствуется, что вычислять такую функцию можно достаточно быстро; тем более, я что-то такое уже где-то видел... :) К тому же, для измерения расстояний можно использовать угол между векторами.
Что до карт Кохонена, то здесь ещё один довод в их пользу пришел в голову. Если снижать размерность выборки с помощью метода главных компонент (в его классической постановке), то в результате будет найден базис, в разложении по которому выборка выглядит наиболее выгодно и лучше всего кластеризуется. Однако, учитывая заявление о "биениях по всего двум координатам" (честно говоря, не понял, что это означает :) ), можно предположить, что у каждого кластера могут быть свои оптимальные ГК (i.e. кластеры ориентированы хаотично). Так вот карты Кохонена, по общим соображениям, в этом случае извлекут, в некотором смысле, больше информации... Правда, я нигде разбора этого вопроса не встречал (может быть потому, что вообще ИНС и ГА недолюбливаю, да и на чистую кластеризацию особого внимания никогда не обращал, сосредотачиваясь вместо этого на вопросах классификации и распознавания). :)
Ах да, чуть не забыл, ещё один маленький совет по ГА. Хромосомы неплохо бы было представить битовыми массивами в кодах Грея. При этом "сила" мутаций будет пропорциональна изменению решения (с одной стороны, мелкие мутации не будут приводить к непредсказуемым скачкам центроидов, с другой же -- для тонкой подстройки центроидов не понадобятся значительные мутации). Хотя, наверное, это лишнее...
Ещё я где-то слышал, что результат ГА-кластеризации хорошо "шлифуется" методом k-средних. То есть, в этом случае, кластеризация выполняется преимущественно все-таки именно методом k-средних, но, так как этот метод слишком чувствителен к начальному решению, то ГА здесь используется для выбора этого налучшего начального приближения. Даже так...
Без экспериментов лучший подход не выбрать... :) А пока выводы таковы: k-means -- не самый качественный, но, думаю, очень быстрый метод, который, однако, может потребовать предварительной подготовки данных; самоорганизующиеся карты Кохонена тормозят, но, также как и k-means, хорошо работают с большими и многомерными выборками, правда при этом саму кластеризацию почти не производят и требуют дополнительного алгоритма для интерпретации результатов, неплохих результатов; ГА хорошо себя чувствуют в малых размерностях и малых объемах информации, полезны для пред- и/или постобработки данных. Среди гибридов можно упомянуть алгоритмы FGKA и IGKA, которые описаны соответственно в Yi Lu et al., FGKA: A Fast Genetic K-means Algorithm, и в Yi Lu et al., Incremental Genetic K-means Algorithm and its Application in Gene Expression Data Analysis.
Существует ещё класс инкрементных алгоримов кластеризации, специально заточенных для работы в изменяющихся условиях (т.е. при добавлении/удалении точек данных). В основном, эти алгоритмы эксплуатируют методики классификации: когда появляется новая точка данных, такой алгоритм относит её к какому-нибудь из имеющихся классов (кластеров) или создает новый (возможно склеивая некоторые из существующих). Причем все спроектировано так, чтобы добавлять/удалять точки можно было с минимальными затратами времени/памяти. Это очень эффективные и экономичные алгоритмы кластеризации, но здесь есть один подвох... Разбиение сильно зависит от порядка поступления данных и это почти непоправимо. :) Один двольно старый метод описан в F.Can, Incremental Clustering for Dynamic Information Processing. Ещё можно глянуть M.Charikar et al., Incremental Clustering and Dynamic Information Retrieval.
Не могу не упомянуть довольно забавный и в тоже время широко используемый подход к кластеризации. Идея заключается в том, чтобы оценить распределение плотности точек данных по пространству и к полученному полю применить пороговый фильтр, после чего произвести заливку кластеров краской. Ну примерно так, есть пути для оптимизации. :) Типичные представители: алгоритмы DBSCAN (выращивающий кластеры, добавляя в них достаточно близкие точки) и OPTICS (суть усовершенствование предыдущего), их описания можно найти в Ester et al., A Density-based Algorithm for Discovering Clusters in Large Spatial Databases with Noise и в M.Ankerst et al., OPTICS: Ordering Points To Identify the Clustering Structure.